- scikit 学习树 - Python (1)
- python中的scikit学习库(1)
- scikit 学习树 - Python 代码示例
- python代码示例中的scikit学习库
- Scikit学习教程(1)
- Scikit学习教程
- Scikit学习-简介(1)
- Scikit学习-简介
- 讨论Scikit学习(1)
- 讨论Scikit学习
- 机器学习-Scikit学习算法
- 机器学习-Scikit学习算法(1)
- scikit 学习岭回归 - Python (1)
- Scikit学习-KNN学习
- Scikit学习-KNN学习(1)
- scikit 学习岭回归 - Python 代码示例
- scikit 学习拆分数据集 - Python (1)
- Scikit学习-决策树
- Scikit学习-决策树(1)
- Scikit学习-有用的资源
- Scikit学习-有用的资源(1)
- Scikit学习-增强方法
- Scikit学习-增强方法(1)
- Scikit学习-约定
- Scikit学习-约定(1)
- Scikit学习-聚类方法
- Scikit学习-聚类方法(1)
- Scikit学习-异常检测
- Scikit学习-异常检测(1)
📅 最后修改于: 2020-12-10 05:47:56 🧑 作者: Mango
在本章中,我们将学习Estimator API (应用程序编程接口)。让我们首先了解什么是Estimator API。
什么是估算器API
它是Scikit-learn实现的主要API之一。它为各种ML应用程序提供了一致的接口,这就是Scikit-Learn中所有机器学习算法都是通过Estimator API实现的原因。从数据中学习(拟合数据)的对象是估计量。它可以与分类,回归,聚类的任何算法一起使用,甚至可以与从原始数据中提取有用特征的转换器一起使用。
为了拟合数据,所有估计器对象公开一个fit方法,该方法采用如下所示的数据集:
estimator.fit(data)
接下来,当通过相应的属性实例化估算器时,可以如下设置估算器的所有参数。
estimator = Estimator (param1=1, param2=2)
estimator.param1
上面的输出为1。
将数据与估算器拟合后,即可根据手头的数据估算参数。现在,所有估计的参数将成为估计器对象的属性,以下划线结尾,如下所示:
estimator.estimated_param_
估算器API的使用
估计器的主要用途如下-
模型的估计和解码
估计器对象用于模型的估计和解码。此外,该模型被估计为以下确定性函数-
-
对象构造中提供的参数。
-
如果估计器的random_state参数设置为none,则为全局随机状态(numpy.random)。
-
传递给最新调用fit,fit_transform或fit_predict的任何数据。
-
在对partial_fit的调用序列中传递的任何数据。
将非矩形数据表示映射到矩形数据
它将非矩形数据表示形式映射为矩形数据。简而言之,它接受输入,其中每个样本都不表示为固定长度的数组状对象,并为每个样本产生特征的数组状对象。
核心样本与外围样本之间的区别
它使用以下方法对核心样本和外围样本之间的区别进行建模-
-
适合
-
fit_predict如果是转导的
-
预测是否归纳
指导原则
在设计Scikit-Learn API时,请牢记以下指导原则-
一致性
该原则指出,所有对象应该共享从一组有限的方法中提取的公共接口。文档也应保持一致。
受限的对象层次
这个指导原则说-
-
算法应由Python类表示
-
数据集应以标准格式表示,例如NumPy数组,Pandas DataFrames,SciPy稀疏矩阵。
-
参数名称应使用标准Python字符串。
组成
众所周知,机器学习算法可以表示为许多基本算法的序列。 Scikit-learn会在需要时使用这些基本算法。
合理的默认值
根据此原理,只要ML模型需要用户指定的参数,Scikit-learn库就会定义适当的默认值。
检查
根据此指导原则,每个指定的参数值都公开为公共属性。
使用Estimator API的步骤
以下是使用Scikit-Learn估计器API的步骤-
步骤1:选择模型类别
在第一步中,我们需要选择一类模型。可以通过从Scikit-learn导入适当的Estimator类来完成。
步骤2:选择模型超参数
在这一步中,我们需要选择类模型超参数。可以通过用所需的值实例化类来完成。
步骤3:整理资料
接下来,我们需要将数据排列到特征矩阵(X)和目标向量(y)中。
步骤4:模型拟合
现在,我们需要使模型适合您的数据。可以通过调用模型实例的fit()方法来完成。
步骤5:应用模型
拟合模型后,我们可以将其应用于新数据。对于监督学习,请使用predict()方法来预测未知数据的标签。对于无监督学习,请使用predict()或transform()推断数据的属性。
监督学习的例子
在此,作为此过程的示例,我们以将线拟合到(x,y)数据的简单情况为例,即简单线性回归。
首先,我们需要加载数据集,我们使用虹膜数据集-
例
import seaborn as sns
iris = sns.load_dataset('iris')
X_iris = iris.drop('species', axis = 1)
X_iris.shape
输出
(150, 4)
例
y_iris = iris['species']
y_iris.shape
输出
(150,)
例
现在,对于此回归示例,我们将使用以下样本数据-
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
rng = np.random.RandomState(35)
x = 10*rng.rand(40)
y = 2*x-1+rng.randn(40)
plt.scatter(x,y);
输出
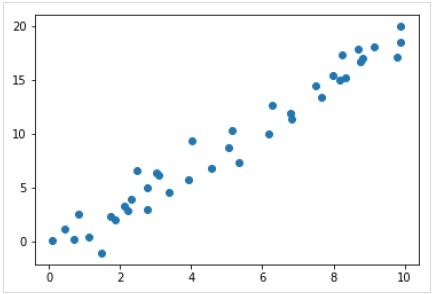
因此,对于线性回归示例,我们具有上述数据。
现在,利用这些数据,我们可以应用上述步骤。
选择一种型号
在这里,要计算一个简单的线性回归模型,我们需要导入线性回归类,如下所示:
from sklearn.linear_model import LinearRegression
选择模型超参数
选择一类模型后,我们需要做出一些重要的选择,这些选择通常表示为超参数,或者是在模型适合数据之前必须设置的参数。在这里,对于线性回归的示例,我们想通过使用fit_intercept超参数来拟合截距,如下所示:
例
model = LinearRegression(fit_intercept = True)
model
输出
LinearRegression(copy_X = True, fit_intercept = True, n_jobs = None, normalize = False)
整理数据
现在,我们知道我们的目标变量y的格式正确,即长度为1-D的n_samples数组。但是,我们需要调整特征矩阵X的形状,使其成为大小为[n_samples,n_features]的矩阵。它可以做到如下-
例
X = x[:, np.newaxis]
X.shape
输出
(40, 1)
模型拟合
一旦我们整理了数据,就该对模型进行拟合了,即将模型应用于数据。这可以借助fit()方法完成,如下所示:
例
model.fit(X, y)
输出
LinearRegression(copy_X = True, fit_intercept = True, n_jobs = None,normalize = False)
在Scikit-learn中, fit()进程带有一些下划线。
对于此示例,以下参数显示了数据的简单线性拟合的斜率-
例
model.coef_
输出
array([1.99839352])
以下参数表示对数据的简单线性拟合的截距-
例
model.intercept_
输出
-0.9895459457775022
将模型应用于新数据
训练模型后,我们可以将其应用于新数据。监督式机器学习的主要任务是根据不是训练集一部分的新数据评估模型。可以借助预测()方法完成以下操作-
例
xfit = np.linspace(-1, 11)
Xfit = xfit[:, np.newaxis]
yfit = model.predict(Xfit)
plt.scatter(x, y)
plt.plot(xfit, yfit);
输出
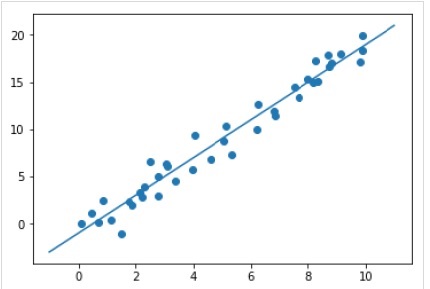
完整的工作/可执行示例
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
iris = sns.load_dataset('iris')
X_iris = iris.drop('species', axis = 1)
X_iris.shape
y_iris = iris['species']
y_iris.shape
rng = np.random.RandomState(35)
x = 10*rng.rand(40)
y = 2*x-1+rng.randn(40)
plt.scatter(x,y);
from sklearn.linear_model import LinearRegression
model = LinearRegression(fit_intercept=True)
model
X = x[:, np.newaxis]
X.shape
model.fit(X, y)
model.coef_
model.intercept_
xfit = np.linspace(-1, 11)
Xfit = xfit[:, np.newaxis]
yfit = model.predict(Xfit)
plt.scatter(x, y)
plt.plot(xfit, yfit);
无监督学习示例
在此,作为此过程的示例,我们以降低Iris数据集的维数为例,以便我们可以更轻松地对其进行可视化。对于此示例,我们将使用主成分分析(PCA),一种快速线性降维技术。
像上面给出的示例一样,我们可以从虹膜数据集中加载并绘制随机数据。之后,我们可以按照以下步骤操作-
选择一种型号
from sklearn.decomposition import PCA
选择模型超参数
例
model = PCA(n_components=2)
model
输出
PCA(copy = True, iterated_power = 'auto', n_components = 2, random_state = None,
svd_solver = 'auto', tol = 0.0, whiten = False)
模型拟合
例
model.fit(X_iris)
输出
PCA(copy = True, iterated_power = 'auto', n_components = 2, random_state = None,
svd_solver = 'auto', tol = 0.0, whiten = False)
将数据转换为二维
例
X_2D = model.transform(X_iris)
现在,我们可以将结果绘制如下:
输出
iris['PCA1'] = X_2D[:, 0]
iris['PCA2'] = X_2D[:, 1]
sns.lmplot("PCA1", "PCA2", hue = 'species', data = iris, fit_reg = False);
输出
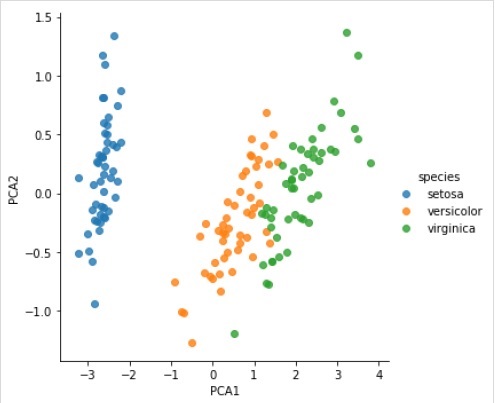
完整的工作/可执行示例
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
iris = sns.load_dataset('iris')
X_iris = iris.drop('species', axis = 1)
X_iris.shape
y_iris = iris['species']
y_iris.shape
rng = np.random.RandomState(35)
x = 10*rng.rand(40)
y = 2*x-1+rng.randn(40)
plt.scatter(x,y);
from sklearn.decomposition import PCA
model = PCA(n_components=2)
model
model.fit(X_iris)
X_2D = model.transform(X_iris)
iris['PCA1'] = X_2D[:, 0]
iris['PCA2'] = X_2D[:, 1]
sns.lmplot("PCA1", "PCA2", hue='species', data=iris, fit_reg=False);