Python中的多维数据分析
多维数据分析是对数据的信息分析,它考虑了许多关系。让我们阐明一些用于使用Python编写的开源库分析多维/多变量数据的基本技术。
从这里找到用于说明的数据链接。
以下代码用于从 zoo_data.csv 读取 2D 表格数据。
Python3
import pandas as pd
zoo_data = pd.read_csv("zoo_data.csv", encoding = 'utf-8',
index_col = ["animal_name"])
# print first 5 rows of zoo data
print(zoo_data.head())Python3
# from sklearn.cluster import KMeans
clusters = 7
kmeans = KMeans(n_clusters = clusters)
kmeans.fit(zoo_data)
print(kmeans.labels_)Python3
# from sklearn.decomposition import PCA
pca = PCA(3)
pca.fit(zoo_data)
pca_data = pd.DataFrame(pca.transform(zoo_data))
print(pca_data.head())Python3
from matplotlib import colors as mcolors
import math
''' Generating different colors in ascending order
of their hsv values '''
colors = list(zip(*sorted((
tuple(mcolors.rgb_to_hsv(
mcolors.to_rgba(color)[:3])), name)
for name, color in dict(
mcolors.BASE_COLORS, **mcolors.CSS4_COLORS
).items())))[1]
# number of steps to taken generate n(clusters) colors
skips = math.floor(len(colors[5 : -5])/clusters)
cluster_colors = colors[5 : -5 : skips]Python3
from mpl_toolkits.mplot3d import Axes3D
import matplotlib.pyplot as plt
fig = plt.figure()
ax = fig.add_subplot(111, projection = '3d')
ax.scatter(pca_data[0], pca_data[1], pca_data[2],
c = list(map(lambda label : cluster_colors[label],
kmeans.labels_)))
str_labels = list(map(lambda label:'% s' % label, kmeans.labels_))
list(map(lambda data1, data2, data3, str_label:
ax.text(data1, data2, data3, s = str_label, size = 16.5,
zorder = 20, color = 'k'), pca_data[0], pca_data[1],
pca_data[2], str_labels))
plt.show()Python3
import seaborn as sns
# generating correlation heatmap
sns.heatmap(zoo_data.corr(), annot = True)
# posting correlation heatmap to output console
plt.show()Python3
from matplotlib import cm
# generating correlation data
df = zoo_data.corr()
df.index = range(0, len(df))
df.rename(columns = dict(zip(df.columns, df.index)), inplace = True)
df = df.astype(object)
''' Generating coordinates with
corresponding correlation values '''
for i in range(0, len(df)):
for j in range(0, len(df)):
if i != j:
df.iloc[i, j] = (i, j, df.iloc[i, j])
else :
df.iloc[i, j] = (i, j, 0)
df_list = []
# flattening dataframe values
for sub_list in df.values:
df_list.extend(sub_list)
# converting list of tuples into trivariate dataframe
plot_df = pd.DataFrame(df_list)
fig = plt.figure()
ax = Axes3D(fig)
# plotting 3D trisurface plot
ax.plot_trisurf(plot_df[0], plot_df[1], plot_df[2],
cmap = cm.jet, linewidth = 0.2)
plt.show()输出:

注意:我们这里的数据类型通常是分类的。本案例研究中用于分类数据分析的技术是非常基本的技术,易于理解、解释和实施。这些包括聚类分析、相关分析、PCA(主成分分析)和 EDA(探索性数据分析)分析。
聚类分析:
由于我们拥有的数据是基于不同类型动物的特征,我们可以使用一些众所周知的聚类技术将动物分为不同的组(集群)或子组,即 KMeans 聚类、DBscan、层次聚类和 KNN(K-Nearest Neighbours)聚类。为简单起见,在这种情况下,KMeans 聚类应该是更好的选择。使用 Kmeans 聚类技术对数据进行聚类可以使用 sklearn 库的 cluster 类的 KMeans 模块实现,如下所示:
Python3
# from sklearn.cluster import KMeans
clusters = 7
kmeans = KMeans(n_clusters = clusters)
kmeans.fit(zoo_data)
print(kmeans.labels_)
输出:

在这里,整体集群惯性为119.70392382759556 。该值存储在 kmeans.inertia_ 变量中。 EDA分析:
为了执行 EDA 分析,我们需要降低多元数据的维数,我们必须对数据进行三元/双元(2D/3D)数据。我们可以使用 PCA(主成分分析)来完成这项任务。
有关更多信息,请参阅 https://www.geeksforgeeks.org/Dimensionity-reduction/
PCA可以使用库sklearn的类分解的PCA模块进行如下:
Python3
# from sklearn.decomposition import PCA
pca = PCA(3)
pca.fit(zoo_data)
pca_data = pd.DataFrame(pca.transform(zoo_data))
print(pca_data.head())
输出:

上面的数据输出代表我们可以执行 EDA 分析的简化三变量 (3D) 数据。
注意: PCA 生成的缩减数据可以间接用于执行各种分析,但不能直接用于人类解释。
散点图是一种 2D/3D 图,有助于分析 2D/3D 数据中的各种聚类。
我们之前生成的 3D 缩减数据的散点图可以绘制如下:
下面的代码是一个Pythonic代码,它生成一个颜色数组(其中颜色的数量大约等于簇的数量),按色调、值和饱和度值的顺序排序。在这里,每种颜色都与单个集群相关联,并将用于将动物表示为 3D 点,同时将其绘制在 3D 绘图/空间中(在本例中为散点图)。
Python3
from matplotlib import colors as mcolors
import math
''' Generating different colors in ascending order
of their hsv values '''
colors = list(zip(*sorted((
tuple(mcolors.rgb_to_hsv(
mcolors.to_rgba(color)[:3])), name)
for name, color in dict(
mcolors.BASE_COLORS, **mcolors.CSS4_COLORS
).items())))[1]
# number of steps to taken generate n(clusters) colors
skips = math.floor(len(colors[5 : -5])/clusters)
cluster_colors = colors[5 : -5 : skips]
下面的代码是一个pythonic代码,它生成一个 3D 散点图,其中每个数据点都有一个与其对应的集群相关的颜色。
Python3
from mpl_toolkits.mplot3d import Axes3D
import matplotlib.pyplot as plt
fig = plt.figure()
ax = fig.add_subplot(111, projection = '3d')
ax.scatter(pca_data[0], pca_data[1], pca_data[2],
c = list(map(lambda label : cluster_colors[label],
kmeans.labels_)))
str_labels = list(map(lambda label:'% s' % label, kmeans.labels_))
list(map(lambda data1, data2, data3, str_label:
ax.text(data1, data2, data3, s = str_label, size = 16.5,
zorder = 20, color = 'k'), pca_data[0], pca_data[1],
pca_data[2], str_labels))
plt.show()
输出:
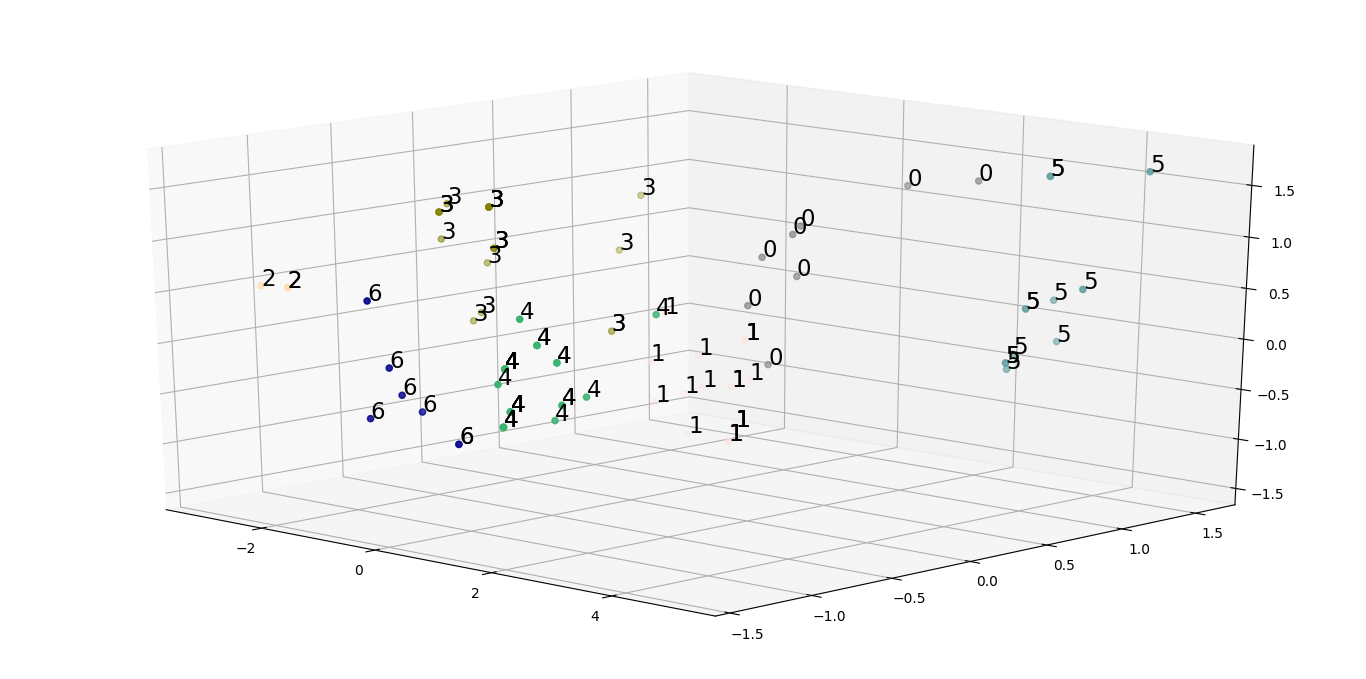
仔细分析散点图可能会导致假设使用初始数据形成的集群没有足够好的解释能力。为了解决这个问题,我们需要将我们的特征集缩减为更有用的特征集,使用这些特征我们可以生成有用的集群。产生这样一组特征的一种方法是进行相关性分析。这可以通过绘制热图和三角形图来完成,如下所示:
Python3
import seaborn as sns
# generating correlation heatmap
sns.heatmap(zoo_data.corr(), annot = True)
# posting correlation heatmap to output console
plt.show()
输出:
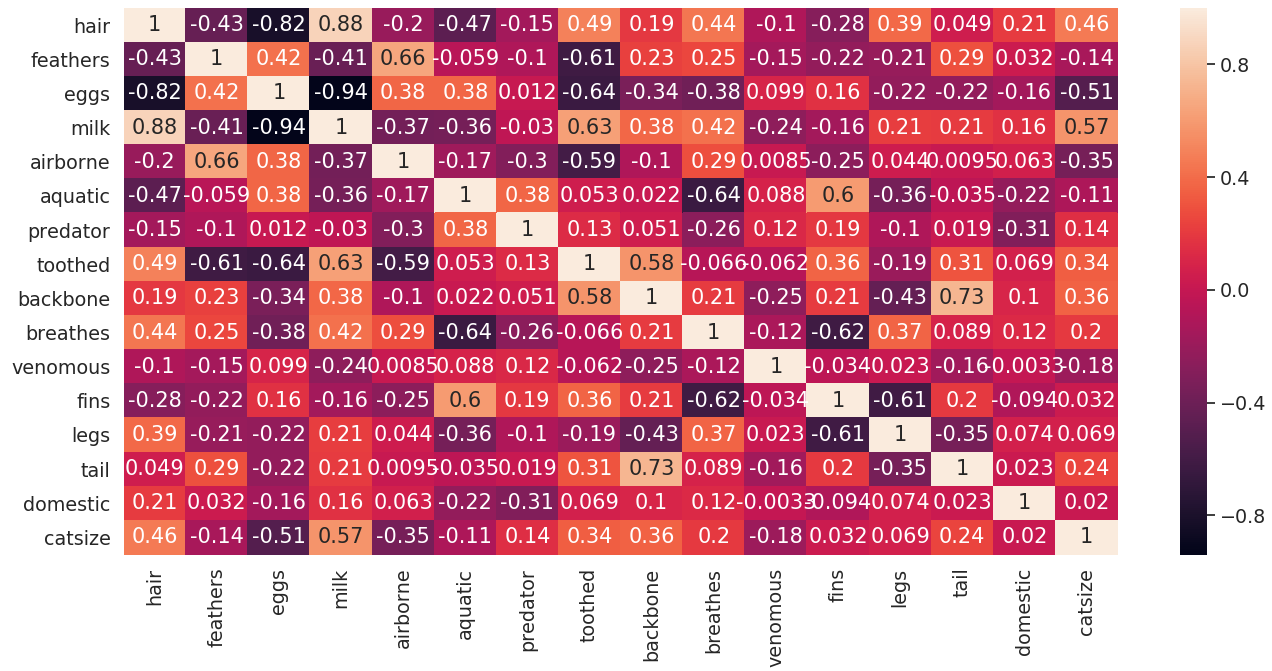
以下代码用于通过制作元组列表来生成相关矩阵的三角图,其中元组包含按动物名称顺序排列的坐标和相关值。
上述解释的伪代码:
# PseudoCode
tuple -> (position_in_dataframe(feature1),
position_in_dataframe(feature2),
correlation(feature1, feature2))为相关矩阵生成三曲面图的代码:
Python3
from matplotlib import cm
# generating correlation data
df = zoo_data.corr()
df.index = range(0, len(df))
df.rename(columns = dict(zip(df.columns, df.index)), inplace = True)
df = df.astype(object)
''' Generating coordinates with
corresponding correlation values '''
for i in range(0, len(df)):
for j in range(0, len(df)):
if i != j:
df.iloc[i, j] = (i, j, df.iloc[i, j])
else :
df.iloc[i, j] = (i, j, 0)
df_list = []
# flattening dataframe values
for sub_list in df.values:
df_list.extend(sub_list)
# converting list of tuples into trivariate dataframe
plot_df = pd.DataFrame(df_list)
fig = plt.figure()
ax = Axes3D(fig)
# plotting 3D trisurface plot
ax.plot_trisurf(plot_df[0], plot_df[1], plot_df[2],
cmap = cm.jet, linewidth = 0.2)
plt.show()
输出:
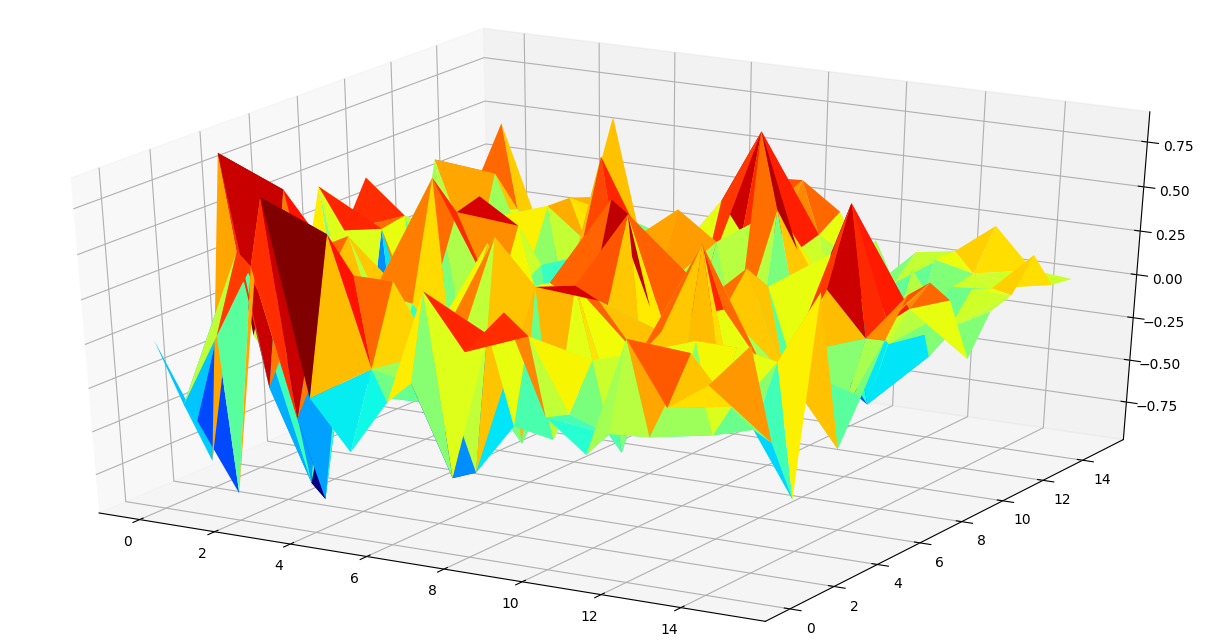
使用热图和三角图,我们可以推断出如何选择一组较小的特征用于执行聚类分析。通常,具有极端相关值的特征对具有很高的解释力,可用于进一步分析。
在这种情况下,查看两个图,我们得出了一个包含 7 个特征的合理列表: [“牛奶”、“鸡蛋”、“头发”、“牙齿”、“羽毛”、“呼吸”、“水生”]
在子集特征集上再次运行聚类分析,我们可以生成散点图,更好地推断如何在不同的群体之间传播不同的动物。
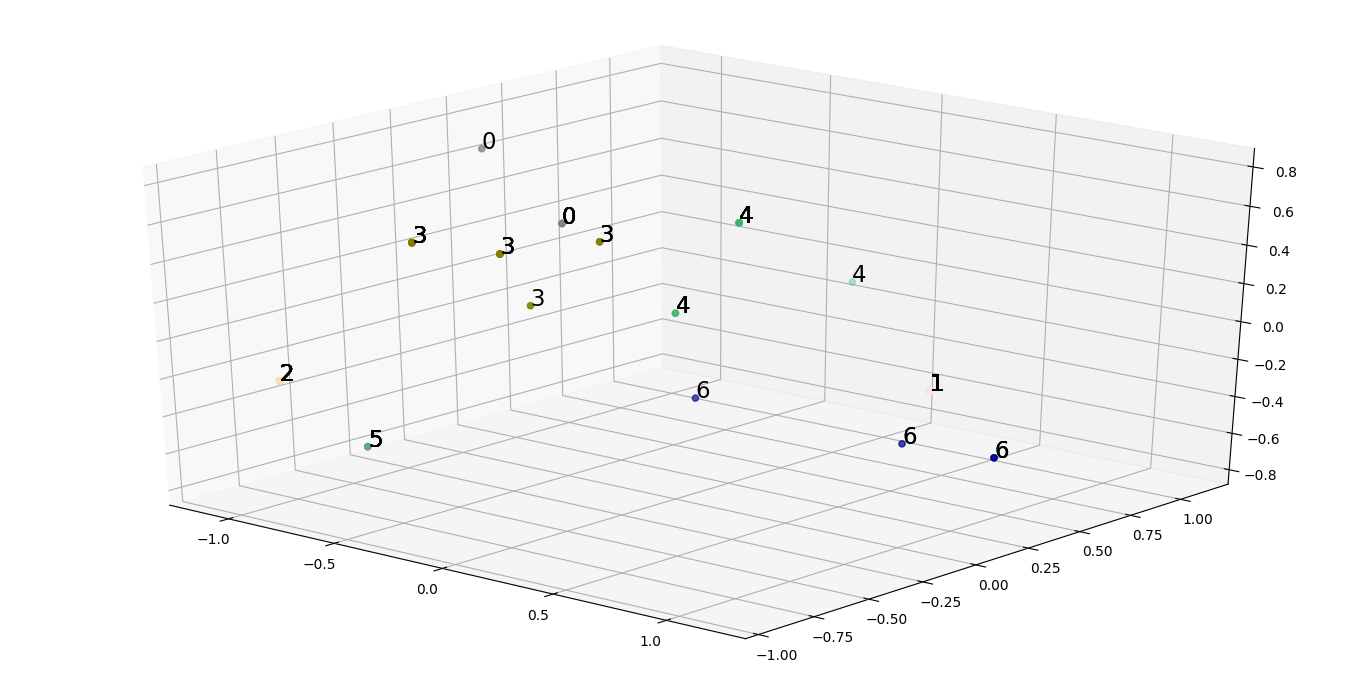
我们观察到整体惯性减少了14.479670329670329 ,这确实比初始惯性小得多。