双支持向量机
先决条件:在 SVM 中分离超平面
支持向量机的拉格朗日乘子方程。其方程可由下式给出:
现在,根据对偶原理,上面的优化问题可以被视为既是原始的(最小化w 和b)或双重(最大化a )。
凸优化的 Slater 条件保证这两个是相等的问题。
要获得 wrt w 和 b 的最小值,这些变量的一阶偏导数必须为 0:
现在,将上述方程代入拉格朗日乘子方程并化简。
在上面的等式中,项
要找到 b,我们还可以使用上面的等式和约束
现在,决策规则可以由下式给出:
注意,我们可以从上面的规则中观察到,拉格朗日乘数只取决于 x i与未知变量 x 的点积。这个点积定义为核函数,用K表示
现在,对于线性不可分的情况,对偶方程变为:
在这里,我们添加了一个常量C,它是必需的,原因如下:
- 它阻止了价值
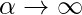
- 它还可以防止模型过度拟合,这意味着可以接受一些错误分类。
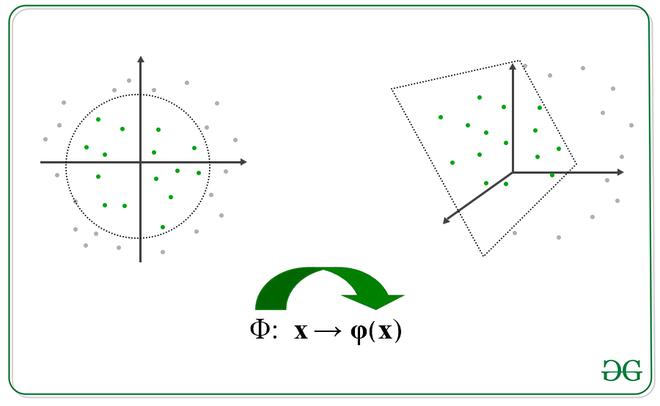
描述转换的图像
我们将变换应用到另一个空间,如下。注意,我们不需要专门计算转换函数,我们只需要找到它们的点积就可以得到核函数,但是,这个转换函数可以很容易地建立。
在哪里,
很多时候数据可以由更高维度的超平面分隔的直觉。让我们更详细地看一下:
假设我们有一个仅包含 1 个自变量和 1 个因变量的数据集。下图表示数据:
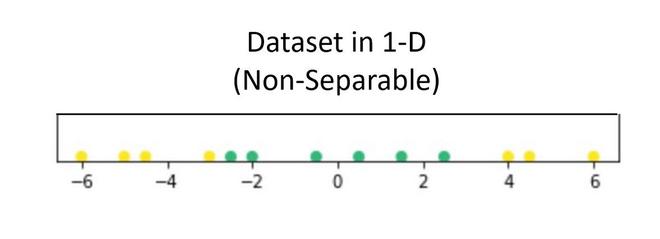
现在,在上面的图中,很难分离清楚地将不同类的数据点分开的一维超平面(点)。但是当通过使用某种转换将其转换为 2d 时,它提供了用于分离类的选项。

在上面的例子中,我们可以看到一条 SVM 线可以清楚地将数据集的两个类分开。
有一些非常常用的著名内核:
- 阶数 =n 的多项式
- 次数最多为 n 的多项式
- 高斯/RBF核
执行
Python3
# code
import numpy as np
import matplotlib.pyplot as plt
from sklearn import svm, datasets
# import some data
cancer = datasets.load_breast_cancer()
X = cancer.data[:,:2]
Y = cancer.target
X.shape, Y.shape
# perform svm with different kernel, here c is the regularizer
h = .02
C=100
lin_svc = svm.LinearSVC(C=C)
svc = svm.SVC(kernel='linear', C=C)
rbf_svc = svm.SVC(kernel='rbf', gamma=0.7, C=C)
poly_svc = svm.SVC(kernel='poly', degree=3, C=C)
# Fit the training dataset.
lin_svc.fit(X, Y)
svc.fit(X, Y)
rbf_svc.fit(X, Y)
poly_svc.fit(X, Y)
# plot the results
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, h),np.arange(y_min, y_max, h))
titles = ['linear kernel',
'LinearSVC (linear kernel)',
'RBF kernel',
'polynomial (degree 3) kernel']
plt.figure(figsize=(10,10))
for i, clf in enumerate((svc, lin_svc,rbf_svc, poly_svc )):
# Plot the decision boundary using the above meshgrid we generated
plt.subplot(2, 2, i + 1)
Z = clf.predict(np.c_[xx.ravel(), yy.ravel()])
# Put the result into a color plot
Z = Z.reshape(xx.shape)
plt.set_cmap(plt.cm.flag_r)
plt.contourf(xx, yy, Z)
# Plot also the training points
plt.scatter(X[:, 0], X[:, 1], c=Y)
plt.title(titles[i])
plt.show()((569, 2), (569,))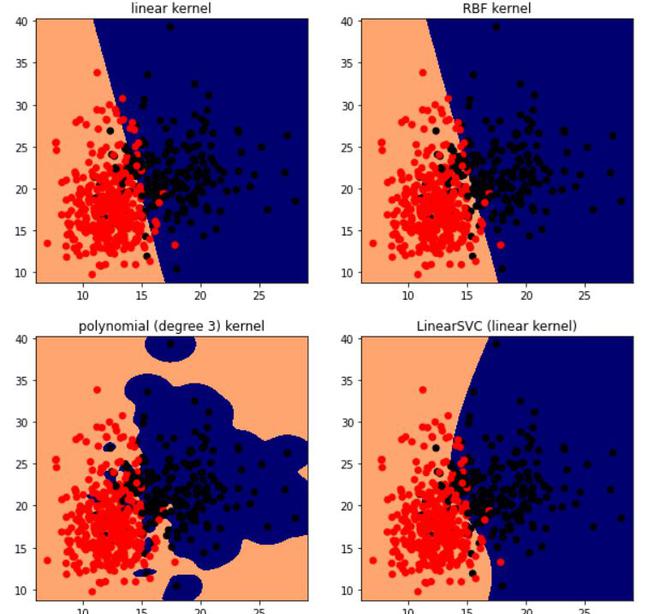
使用不同内核的 SVM。
参考:
- MIT OCW 幻灯片 SVM