- 聚类算法-概述
- 聚类算法-概述(1)
- R 编程中的聚类
- R 编程中的聚类(1)
- Python 中的 K-Means 聚类 - 3 个聚类 - Python (1)
- K均值和分层聚类之间的区别
- K均值和分层聚类之间的区别(1)
- Python 中的 K-Means 聚类 - 3 个聚类 - Python 代码示例
- 不同类型的聚类算法
- 不同类型的聚类算法(1)
- 不同类型的聚类算法(1)
- 不同类型的聚类算法
- k-means 聚类和禁用聚类 - Python 代码示例
- 使用 Weka 进行分层聚类(1)
- 使用 Weka 进行分层聚类
- Julia 中的聚类(1)
- Julia 中的聚类
- Julia 中的聚类
- Julia 中的聚类(1)
- 聚类算法-K-均值算法
- 数据挖掘中的聚类
- 数据挖掘中的聚类(1)
- CURE 聚类和 DBSCAN 聚类的区别
- CURE 聚类和 DBSCAN 聚类的区别(1)
- 机器学习中的 DBSCAN 聚类 |基于密度的聚类
- 机器学习中的 DBSCAN 聚类 |基于密度的聚类(1)
- rabbitmq 聚类 (1)
- K表示聚类–简介
- K表示聚类–简介(1)
📅 最后修改于: 2020-12-10 05:41:15 🧑 作者: Mango
层次聚类简介
分层聚类是另一种无监督的学习算法,用于将具有相似特征的未标记数据点分组在一起。分层聚类算法分为以下两类-
聚集层次算法-在聚集层次算法中,每个数据点都被视为单个群集,然后连续合并或聚集(自下而上)群集对。群集的层次结构表示为树状图或树结构。
分开的分层算法-另一方面,在分开的分层算法中,所有数据点都被视为一个大群集,并且群集过程涉及将(一个自上而下的方法)将一个大群集划分为各种小群集。
执行聚集层次聚类的步骤
我们将解释最常用和最重要的层次聚类,即聚类。执行相同的步骤如下-
-
步骤1-将每个数据点视为单个群集。因此,开始时我们将拥有K个群集。开始时,数据点的数量也将为K。
-
步骤2-现在,在这一步中,我们需要通过连接两个壁橱数据点来形成一个大型集群。这将导致总共K-1个群集。
-
步骤3-现在,要形成更多集群,我们需要加入两个壁橱集群。这将导致总共有K-2个集群。
-
步骤4-现在,要形成一个大集群,请重复上述三个步骤,直到K变为0,即不再有要加入的数据点。
-
步骤5-最后,在制作了一个大集群之后,将根据问题使用树状图将其划分为多个集群。
树状图在聚集层次聚类中的作用
正如我们在最后一步中讨论的那样,一旦大集群形成,树状图就开始发挥作用。根据我们的问题,将使用树状图将群集分为相关数据点的多个群集。通过以下示例可以理解-
例子1
为了理解,让我们从导入所需的库开始,如下所示:
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
接下来,我们将绘制此示例中采用的数据点-
X = np.array([[7,8],[12,20],[17,19],[26,15],[32,37],[87,75],[73,85], [62,80],[73,60],[87,96],])
labels = range(1, 11)
plt.figure(figsize=(10, 7))
plt.subplots_adjust(bottom=0.1)
plt.scatter(X[:,0],X[:,1], label='True Position')
for label, x, y in zip(labels, X[:, 0], X[:, 1]):
plt.annotate(label,xy=(x, y), xytext=(-3, 3),textcoords='offset points', ha='right', va='bottom')
plt.show()
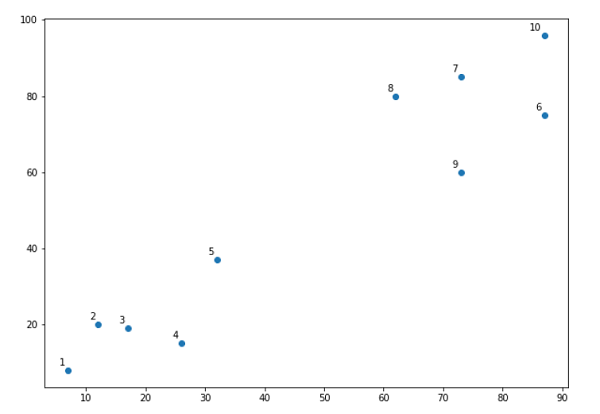
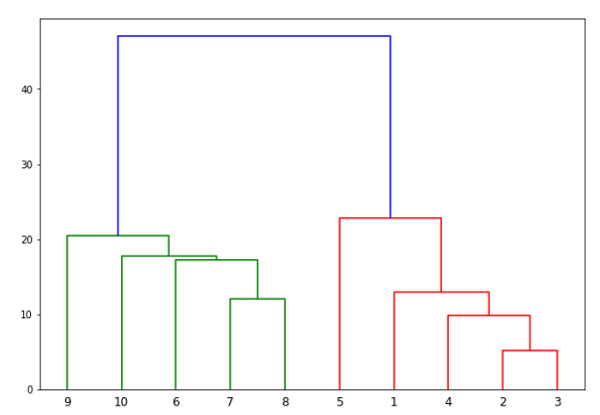
从上图很容易看出,我们在数据点外有两个集群,但在现实世界的数据中,可以有成千上万个集群。接下来,我们将使用Scipy库绘制数据点的树状图-
from scipy.cluster.hierarchy import dendrogram, linkage
from matplotlib import pyplot as plt
linked = linkage(X, 'single')
labelList = range(1, 11)
plt.figure(figsize=(10, 7))
dendrogram(linked, orientation='top',labels=labelList, distance_sort='descending',show_leaf_counts=True)
plt.show()
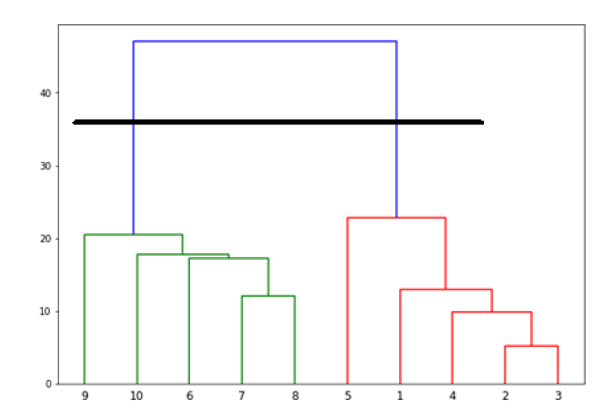
现在,一旦形成大集群,就选择了最长的垂直距离。然后通过一条垂直线绘制一条线,如下图所示。当水平线与蓝线在两个点处相交时,簇的数量将为两个。
接下来,我们需要导入用于聚类的类,并调用其fit_predict方法以预测聚类。我们正在导入sklearn.cluster库的AgglomerativeClustering类-
from sklearn.cluster import AgglomerativeClustering
cluster = AgglomerativeClustering(n_clusters=2, affinity='euclidean', linkage='ward')
cluster.fit_predict(X)
接下来,在以下代码的帮助下绘制群集-
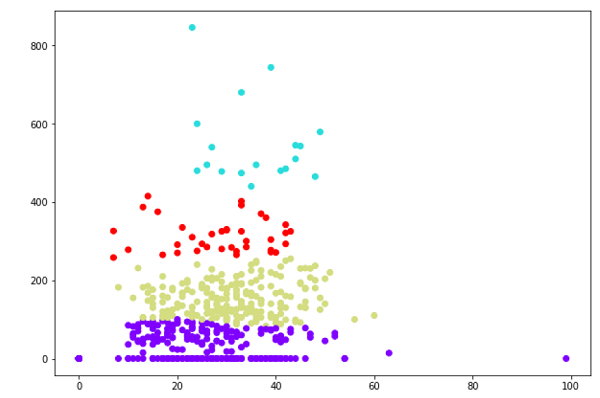
plt.scatter(X[:,0],X[:,1], c=cluster.labels_, cmap='rainbow')
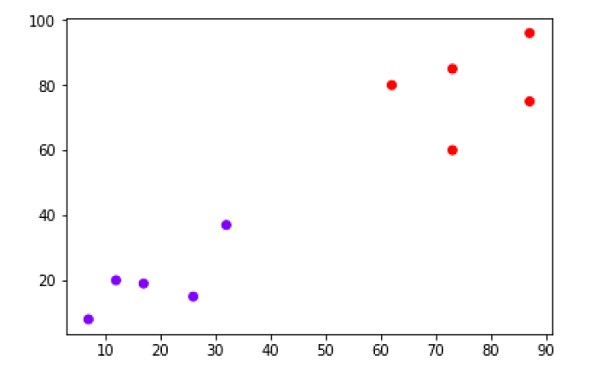
上图显示了来自我们数据点的两个群集。
例2
正如我们从上面讨论的简单示例理解树状图的概念一样,让我们转到另一个示例,在该示例中,我们将使用分层聚类在Pima Indian Diabetes Dataset中创建数据点的聚类-
import matplotlib.pyplot as plt
import pandas as pd
%matplotlib inline
import numpy as np
from pandas import read_csv
path = r"C:\pima-indians-diabetes.csv"
headernames = ['preg', 'plas', 'pres', 'skin', 'test', 'mass', 'pedi', 'age', 'class']
data = read_csv(path, names=headernames)
array = data.values
X = array[:,0:8]
Y = array[:,8]
data.shape
(768, 9)
data.head()
| slno. | preg | Plas | Pres | skin | test | mass | pedi | age | class |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 6 | 148 | 72 | 35 | 0 | 33.6 | 0.627 | 50 | 1 |
| 1 | 1 | 85 | 66 | 29 | 0 | 26.6 | 0.351 | 31 | 0 |
| 2 | 8 | 183 | 64 | 0 | 0 | 23.3 | 0.672 | 32 | 1 |
| 3 | 1 | 89 | 66 | 23 | 94 | 28.1 | 0.167 | 21 | 0 |
| 4 | 0 | 137 | 40 | 35 | 168 | 43.1 | 2.288 | 33 | 1 |
patient_data = data.iloc[:, 3:5].values
import scipy.cluster.hierarchy as shc
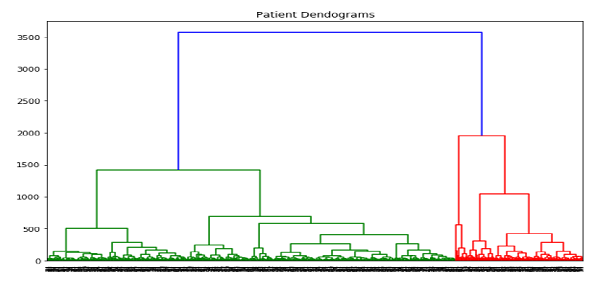
plt.figure(figsize=(10, 7))
plt.title("Patient Dendograms")
dend = shc.dendrogram(shc.linkage(data, method='ward'))
from sklearn.cluster import AgglomerativeClustering
cluster = AgglomerativeClustering(n_clusters=4, affinity='euclidean', linkage='ward')
cluster.fit_predict(patient_data)
plt.figure(figsize=(10, 7))
plt.scatter(patient_data[:,0], patient_data[:,1], c=cluster.labels_, cmap='rainbow')