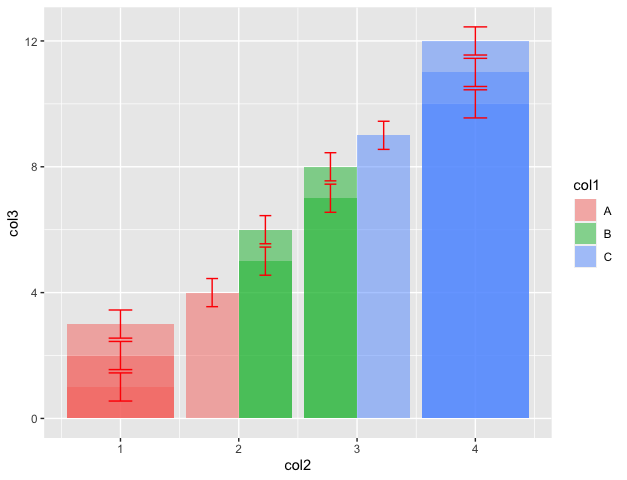
R中带有误差线的分组条形图
在本文中,我们将了解如何在 R 编程语言中创建带有误差线的分组条形图。
可以使用 data.frame() 方法在 R 工作空间中创建数据框。 tidyverse 包被安装并加载到工作空间中,以执行数据突变和操作。
可以使用以下命令将包合并到工作空间中:
install.packages("tidyverse")声明的数据帧使用管道运算符进行大量操作。最初,group_by 方法用于隔离不同组中的数据。它将数据分组的列作为参数。
Syntax: group_by (col-name)
Arguments :
- col-name – The column to group data by
然后可以通过执行将所需列的标准偏差值除以其长度的数学计算来添加临时列。使用 sd() 方法计算标准偏差。长度可以通过 length() 方法计算。这两种方法都将列名作为参数。可以使用 mutate() 方法将该列附加到数据框中。
mutate (new-col-name = func())R 中的 ggplot 方法用于使用指定的数据框进行图形可视化。它用于实例化 ggplot 对象。可以为绘图对象创建美学映射,以分别确定 x 轴和 y 轴之间的关系。可以将其他组件添加到创建的 ggplot 对象中。
Syntax: ggplot(data = NULL, mapping = aes(), fill = )
Arguments :
- data – Default dataset to use for plot.
- mapping – List of aesthetic mappings to use for plot.
可以使用各种方法将几何图形添加到绘图中。 R 中的 geom_line() 方法可用于在绘制的图中添加图形线。它作为组件添加到现有绘图中。审美映射还可以包含颜色属性,这些属性根据不同的数据帧进行不同的分配。
geom_bar() 方法用于构造与每组中的案例数成比例的条形高度。
Syntax: geom_bar ( width, stat)
Arguments :
- width – Bar width
geom_errorbar() 方法用于向图中添加误差线。
Syntax: geom_errorbar(mapping = NULL, data = NULL, stat = “identity”, position = “identity”, …)
Arguments :
- mapping – The aesthetic mapping, usually constructed with aes or aes_string.
- stat – The statistical transformation to use on the data for this layer.
- position – The position adjustment to use for overlapping points on this layer
下面是实现:
R
# importing the required library
library(tidyverse)
data_frame <- data.frame(stringsAsFactors=FALSE,
col1 = c(rep(LETTERS[1:3],each=4)),
col2 = c(rep(1:4,each=3)),
col3 = c(1:12))
print("original dataframe")
print(data_frame)
# computing the length of col3
len <- length(col3)
# plotting the data
data_frame %>%
# grouping by col2
group_by(col2) %>%
# adding a temporary column
mutate(temp_col = sd(col3)/sqrt(len)) %>%
ggplot(aes(x = col2, y = col3, fill = col1)) +
geom_bar(stat="identity", alpha=0.5,
position=position_dodge()) +
# adding error bar
geom_errorbar(aes(ymin=col3-temp_col, ymax=col3+temp_col),
width=.2, colour="red",
position=position_dodge(.9))输出
[1] "original dataframe"
> print(data_frame)
col1 col2 col3
1 A 1 1
2 A 1 2
3 A 1 3
4 A 2 4
5 B 2 5
6 B 2 6
7 B 3 7
8 B 3 8
9 C 3 9
10 C 4 10
11 C 4 11
12 C 4 12