如何用 R 制作 PCA 图
R 编程中的主成分分析(PCA)是对所有现有属性的线性成分的分析。主成分是数据集中原始预测变量的线性组合(正交变换)。它是 EDA(探索性数据分析)的一项有用技术,可让您更好地可视化具有许多变量的数据集中存在的变化。
它最适用于数值数据值。在这个过程中,数据的主成分被计算出来,用于对数据进行基础的改变,有时只使用前几个主成分而忽略其余部分。可以把PCA看作是一种基于一定数据空间的数据线性变换。此变换将数据拟合到坐标系中,其中在第一个坐标上发现最显着的方差,并且每个后续坐标都与最后一个坐标正交,并且方差比前一个坐标小。
R中的PCA图
我们将研究内置于 R 中的“鸢尾花”数据集。它是一个多变量数据集,包含来自三种鸢尾花(山鸢尾、维吉尼亚鸢尾和杂色鸢尾)中每一种的 50 个样本的数据。
R
# structure of the iris
# dataset
str(iris)
# print the iris dataset
head(iris)R
iris.pca <- prcomp(iris[,c(1:4)],
center = TRUE,
scale. = TRUE)
# summary of the
# prcomp object
summary(iris.pca)R
# structure of the pca object
str(iris.pca)R
# loading library
library(ggfortify)
iris.pca.plot <- autoplot(iris.pca,
data = iris,
colour = 'Species')
iris.pca.plotR
biplot.iris.pca <- biplot(iris.pca)
biplot.iris.pcaR
plot.iris.pca <- plot(iris.pca, type="l")
plot.iris.pca输出:

如前所述,PCA 最适用于数值数据,我们将忽略分类变量 Species。我们现在剩下一个 4 列 150 行的矩阵,我们将通过prcomp()函数进行主成分分析。该函数将结果作为“prcomp”类的对象返回。我们将输出分配给名为 iris.pca 的变量。
电阻
iris.pca <- prcomp(iris[,c(1:4)],
center = TRUE,
scale. = TRUE)
# summary of the
# prcomp object
summary(iris.pca)
输出:
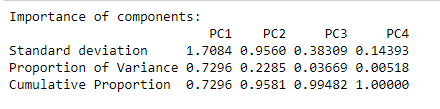
在这里,我们得到了名为 PC1-4 的四个主要组件。这些中的每一个都解释了数据集中总变异的百分比。例如,PC1 解释了将近 72% 的总方差,即大约四分之三的数据集信息可以由一个主成分封装。 PC2 解释了 22% 等等。
让我们看一下如此形成的 PCA 对象的结构。
电阻
# structure of the pca object
str(iris.pca)
输出:

绘制主成分分析
在谈论绘制 PCA 时,我们通常指的是前两个主成分 PC1 和 PC2 的散点图。这些图揭示了数据的特征,例如非线性和偏离正态性。 PC1 和 PC2 对每个样本向量进行评估并绘制。
“ggfortify 包”的autoplot()函数轻松地在 R 中绘制 PCA。
电阻
# loading library
library(ggfortify)
iris.pca.plot <- autoplot(iris.pca,
data = iris,
colour = 'Species')
iris.pca.plot
输出:
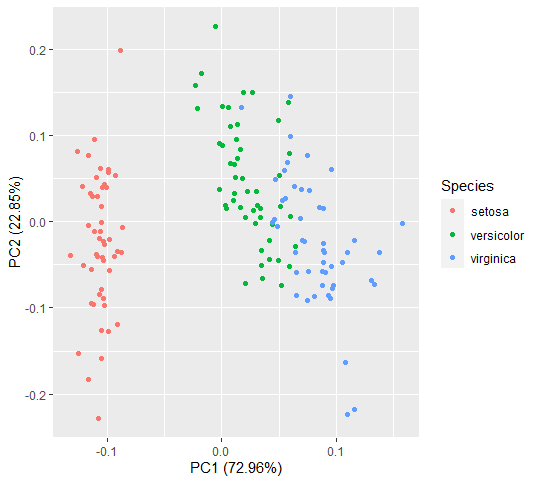
为了更好地理解特征的线性变换, biplot()函数也可用于绘制 PCA。
电阻
biplot.iris.pca <- biplot(iris.pca)
biplot.iris.pca
输出:

双标图的 X 轴代表第一主成分,其中花瓣长度和花瓣宽度组合并转换为具有萼片长度和萼片宽度的某些部分的 PC1。而萼片长度和萼片宽度的垂直部分形成第二主成分。
为了确定在执行 PCA 后可以证明的理想特征,可以使用 plot()函数绘制预压缩对象。
电阻
plot.iris.pca <- plot(iris.pca, type="l")
plot.iris.pca
输出:

在 screeplot 中,“臂弯”代表累积贡献的减少。上图显示了第二个主成分处的弯曲。