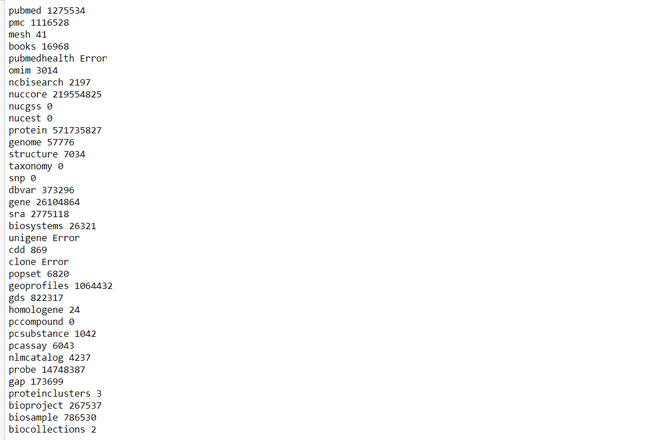Biopython——Entrez数据库搜索操作
NCBI 提供了一个名为 Entrez 的在线搜索系统。这提供了对广泛的分子生物学数据库的访问,它还提供了一个集成的全局查询系统,支持布尔运算符和字段搜索。结果从所有数据库返回,其中包含来自每个数据库的点击次数、原始数据库链接等信息。
使用的功能
Biopython Entrez 配备了两种方法来对数据库执行搜索操作:
- Biopython 有一个名为 esearch ()的 Entrez 特定方法来搜索任何一个 Entrez 数据库。它接受位置参数数据库和我们要搜索的术语。如果分配了错误的数据库,则会引发错误。
句法:
Bio.Entrez.esearch(database, term)
- 要在所有数据库中搜索任何查询,请使用egquery()方法。它类似于Entrez.esearch()方法,除了它只接受 term 参数跳过数据库参数。
句法:
Bio.Entrez.egquery(term)
方法
- 导入所需的模块。
- 设置您的电子邮件以识别谁与数据库连接。
- 设置Entrez工具参数,默认为Biopython。
- 使用上面提供的任何方法和适当的参数。
- 返回的数据将是 XML 格式,因此要在Python对象中获取此数据,请使用Entrez.read()方法读取对象
- 阅读提供的信息。
下面给出了使用这两种方法的实现:
示例 1:使用 esearch()
Python3
# Import libraries
from Bio import Entrez
# Setting email
Entrez.email = 'jeetesh1@yopmail.com'
# Setting Entrez tool parameter
Entrez.tool = 'Demoscript'
# Searching for database
info = Entrez.esearch(db="nucleotide", term="genome")
# reading records
record = Entrez.read(info)
# Showing records
print(record)Python3
# Import libraries
from Bio import Entrez
# Setting email
Entrez.email = 'jeetesh1@yopmail.com'
# Setting Entrez tool parameter
Entrez.tool = 'Demoscript'
# Searching for database
info = Entrez.egquery(term="genome")
record = Entrez.read(info)
for row in record["eGQueryResult"]:
print(row["DbName"], row["Count"])输出:

示例 2:使用 egquery()
蟒蛇3
# Import libraries
from Bio import Entrez
# Setting email
Entrez.email = 'jeetesh1@yopmail.com'
# Setting Entrez tool parameter
Entrez.tool = 'Demoscript'
# Searching for database
info = Entrez.egquery(term="genome")
record = Entrez.read(info)
for row in record["eGQueryResult"]:
print(row["DbName"], row["Count"])
输出 :