正则化判别分析
正则化判别分析
线性判别分析和 QDA 直接适用于观察数量远大于预测变量数量 n>p 的情况。在这些情况下,它提供了很多优势,例如易于应用(因为我们不必计算每个类的协方差)和对模型假设偏差的鲁棒性。
然而,当 LDA 的使用成为一个严峻的挑战时,例如观察的数量少于微阵列设置等预测变量,因为这里有两个挑战
- 样本协方差矩阵是奇异的,不能求逆。
- 高维数使得直接矩阵运算变得强大,从而阻碍了该方法的适用性。
因此,我们会对LDA和QDA做一些改变,即我们形成一个新的协方差矩阵,结合LDA的协方差矩阵( ![]() ) 和 QDA (
) 和 QDA ( ![]() ) 使用调整参数
) 使用调整参数![]()
但是,某些版本的正则化判别分析使用另一个参数( ![]() ) 与以下等式:
) 与以下等式:
RDA 将 QDA 的单独协方差限制为 LDA 的共同协方差。在预测变量的数量大于训练数据中的样本数量的情况下,这改进了协方差矩阵的估计,从而提高了模型的准确性。
在上面的方程中,方程 \gamma 和 \lambda 都有值 b/w 0 和 1。现在,对于所有四个边界值,它为每个边界值生成一个特殊的方程情况。让我们看看这些特殊情况:
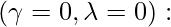 QDA 的协方差,即每个组的个体协方差。
QDA 的协方差,即每个组的个体协方差。 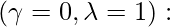 LDA 的协方差,即公共协方差矩阵。
LDA 的协方差,即公共协方差矩阵。 条件独立方差。
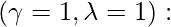 使用欧几里得距离的分类类似于前一种情况,但所有组的方差都相同。
使用欧几里得距离的分类类似于前一种情况,但所有组的方差都相同。
执行
- 在这个实现中,我们将执行正则化判别分析。我们将使用KLAR库,并在它的RDA函数。我们还使用 iris 数据集。
R
# imports
library(tidyverse)
library(MASS)
library(klaR)
data('iris')
# model
# divide the data into train and test
train_test.samples <- iris$Species %>% createDataPartition(p = 0.8, list = FALSE)
train.data <- iris[train_test.samples, ]
test.data <- iris[-train_test.samples, ]
# Data preprocessing
# Normalize the different parameters of dataset and categorical
# variables also includes
preproc.param <- train.data %>%
preProcess(method = c("center", "scale"))
# Transform the data using the estimated parameters
train.transformed <- preproc.param %>% predict(train.data)
test.transformed <- preproc.param %>% predict(test.data)
# define rda models
model = rda(Species ~. , data= train.transformed)
model
# run the model on test data and generate the prediction
predictions <- model %>% predict(test.transformed)
# calculate model accuracy
mean(predictions$class==test.transformed$Species)输出:
Call:
rda(formula = Species ~ ., data = train.transformed)
Regularization parameters:
gamma lambda
0.002619109 0.222244278
Prior probabilities of groups:
setosa versicolor virginica
0.3333333 0.3333333 0.3333333
Misclassification rate:
apparent: 1.667 %
cross-validated: 1.667 %
### accuracy
0.9666667参考:
- 正则化判别分析
- KlaR 文档